挑战常规,是科学家的使命之一。近日,中国科学家毛冠中以共同作者身份发表在 Science 上的新论文,揭示 DNA 结合蛋白得以高效结合 DNA 靶序列的新机制
挑战常规,是科学家的使命之一。近日,中国科学家毛冠中以共同作者身份发表在 Science 上的新论文,揭示 DNA 结合蛋白得以高效结合 DNA 靶序列的新机制。

图 | 毛冠中(来源:毛冠中)
他表示:“该研究得出的实验结果和数学模型,挑战了一直以来教科书所描述的蛋白在基因调控过程中, 精确寻找并结合在基因组特定区域的机制。尽管 DNA 序列是蛋白搜索和蛋白结合的决定性因素,但并非因为特定序列而导致蛋白单次紧密结合的时间更长、以及脱离速度更缓慢,而是因为特定序列更容易被再次结合。”
1 月 27 日,相关论文以《DNA 结合中的序列特异性主要受缔合控制》(Sequence specificity in DNA binding is mainly governed by association)为题,发表在 Science 上 [1]。

图 | 相关论文(来源:Science)
该论文的研究意义在于,针对转录调控的数百种蛋白运作机理,该成果提出了新的方向。这些蛋白直接关系到癌症研究、基于转录调控的新型药物的筛选和开发、遗传病早期发现与治疗、以及农作物性状调整与病虫害抵抗等。
“人生苦短,而蛋白质尤甚”
研究中,毛冠中通过对微观尺度上的单分子 DNA 进行观察与动力学测算,结合蛋白质微阵列批量验证,获悉了以往用宏观生化手段无法观察到的分子缔合与脱离的动力学细节。
基因组本身包含巨量的信息,其中的 DNA 序列长度非常可观。毛冠中指出,纳米级的碱基对组成的单个人体细胞 DNA 链总长度能达快一米,而人体全部 DNA 连接起来并把其拉直,甚至可以超出太阳系的范围。相比之下,这也为数并不算太多的 DNA 结合与搜索功能蛋白提出了巨大难题。

(来源:Science)
人生苦短,而蛋白质尤甚,在一个堪比图书馆容量的信息库里,通常只有几个到十几个碱基对长度的单一指定信息片段,可以决定某项生理功能的激活或关闭,而且给予对应结合蛋白的搜索时间极其有限(微秒到数分钟)。如果想精确分辨每一个碱基对和它的周边组合,即使是 996、007 也永远无法完成工作。因此,必须选择更有效和更有弹性的搜索方式。
这一方式的寻找要从 2020 年说起,毛冠中与所在实验室曾在 Nature 发表了一篇题为《目标搜索过程中的 DNA 表面探测与操纵基因片段跨越》(DNA surface exploration and operator bypassing during target search)的论文 [2]。
在那项研究中,他使用单分子观测手段,观察到了 DNA 结合蛋白 LacI 在序列搜索中的微观往复运动与跳跃,并测量出 LacI 可以略过任意一段 DNA 序列的概率。
LacI 以相当“粗糙”的方式搜索目标区域,并以此达到较高的搜索速度,但这样做的弊端是,LacI 以较高频率略过其原本目标区域,而无法做到每次遇到目标区域就精准结合。不过这样并不会降低总体的结合效率,因为 LacI 在搜寻过程中总是不断冗复访问同样的区域。
(来源:Science)
解决问题的同时又提出了新问题,既然 DNA 序列的具体排列决定了它们是否会成为 DNA 结合蛋白的目标位点,那么在高速搜索时,那些与目标位点相似的序列,是否会成为“迷惑”蛋白的假靶子?从而导致蛋白长时间结合在序列相似的非目标区域?
毕竟在数以百万甚至更多的碱基对排列中,这是无法回避的问题。
以往研究中,科学家在宏观动力学观察中发现,蛋白在其目标位点停留结合的时间,远长于任意非目标序列,因此得出结论,DNA 的具体排序决定了解离速率常数 (koff)。
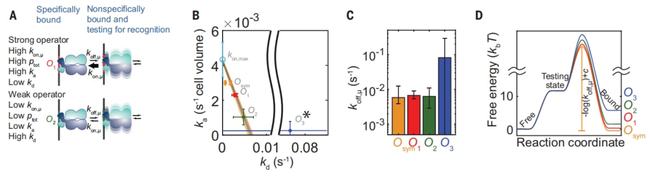
但在毛冠中的最新论文中,他通过单分子共定位观测与计算发现:在微观尺度上,蛋白质与 DNA 的结合时间,并不会因为序列改变而改变。无论目标位点与否,结合上去的 LacI 蛋白都会迅速解离、并继续高速且往复的运动。
那么特异性结合究竟如何实现的?一个简单的数学假设提出了挑战现有教科书理论的反直觉结论,但毛冠中了解缺乏坚实实验数据支持的假设模型是难以获取广泛认可的。于是通过观测结合速率,他发现蛋白在目标序列区域,有着远高于其他区域的结合效率。也就是说,虽然 DNA 结合蛋白会像在任意序列上那样快速解离目标序列,但却能在高速往复搜索运动时,轻易地再次结合在目标序列上,这在宏观测量上的直观体现就是长时间的特异性结合。

(来源:Science)
在分子水平上验证了这个理论后,毛冠中的合作者运用他的共定位观测方法和蛋白质微阵列,对不同 DNA 序列进行批量动力学测量,并建立了相关模型。
如前所述,此次论文的灵感来自于同一项目的前一篇 Nature 论文,在上一篇论文的数据处理中,他发现在不同 DNA 序列的蛋白结合动力学图像中,宏观的结合常数与解离常数(kon & koff)之间存在异常的线性关系。
从通常的看法角度来判断,这两者应当是相对独立的:即在同样的缓冲剂和温度条件下,结合速率与蛋白质浓度,解离速率和 DNA 序列呈现出相关性。因此,毛冠中决定从微观尺度上(单分子实验)对这个反常数据进行观察和测算。
在各方面的支持下,毛冠中克服了显微信号差、蛋白移动结合过快、荧光标记对结合的影响等问题,通过对三个不同的 LacI 天然目标序列的单分子实验测量,得到了微观和宏观尺度上的动力学数据。
他表示:“得益于本实验室精良的单分子荧光显微系统,我们才能在这个看似老生常谈的问题上作出新的突破。对于 DNA 结合蛋白的搜索与精确定位,近年来一直有试图挑战传统宏观测量结论的论文出现,但是受制于观测手段与尺度的局限性,难以提出具有普遍性和系统性、并拥有有力实验结果支持的模型。”
不过,毛冠中也坦言,本次系统虽然在观测尺度上获得突破,但是同样存在局限性。
如果进行大规模不同对象的验证试验,所需要的人力物力将会令实验室难以承受。因此他将把后期批量实验交给合作实验室完成,并由他们最终完成动力学模型细节的建立。
从“异常数据”到完整研究
据介绍,毛冠中祖籍浙江衢州,现家居广东省深圳市,出生于 1985 年。高考后通过面试进入香港科技大学学习生物化学,在出国读硕的空档期,曾在复旦大学微生物系实习半年多。
硕士到博后整个过程都在瑞典王国乌普萨拉大学完成,分别攻读生物技术、分子生物学和生物物理。现在作为高级研究员在乌普萨拉大学塞巴斯蒂安·迪德尔(Sebastian Deindl)教授实验室工作。
毛冠中说:“此前,尚未在上一个实验室完成博士论文和答辩的我,来到塞巴斯蒂安实验室问是否能来这里当博后时,他并没有用各种借口或行政规定搪塞我,仅从专业角度询问我的意向和技能,便决定让我加入。在我的毕业论文因为文法和英语写作的不足而停滞不前时,塞巴斯蒂安自费近万元,请相关专业的资深英语母语教授为我进行指导和修改。”
回顾研究过程,他表示:“在导师塞巴斯蒂安·迪德尔(Sebastian Deindl)教授的有力领导下,让项目从‘异常数据’变成了完整的研究。”
一般来说,某个基因组序列是否能接触到各种转录或调控复合体,由染色体重塑决定。借此可实现 DNA 转录、损伤修复、调控等目的,这与癌症、衰老进程、以及部分遗传病的机理紧密相关,也和农作物、牲畜的性状改变有直接联系。
下一步,毛冠中打算把研究中心移回实验室原本的特长项目上来,前面研究都是基于细菌中的 LacI 蛋白与调控体系,而该实验室主要研究的主要是核小体与染色体重塑。
他计划把已经建立的实验体系用在染色体重塑和真核生物的转录调控上,同时为了解决现有单分子系统的低通量局限性,毛冠中准备结合其他生化、或生物物理手段,打造高通量筛选测量平台。
-End-
参考:
1、Marklund, E., Mao, G., Yuan, J., Zikrin, S., Abdurakhmanov, E., Deindl, S., & Elf, J. (2022). Sequence specificity in DNA binding is mainly governed by association. Science, 375(6579), 442-445.
2、Marklund, E., van Oosten, B., Mao, G., Amselem, E., Kipper, K., Sabantsev, A., ... & Deindl, S. (2020). DNA surface exploration and operator bypassing during target search. Nature, 583(7818), 858-861.
标签: 结合 中国 科学家 揭示 蛋白 序列 新机制 实现 DNA
声明:本文内容来源自网络,文字、图片等素材版权属于原作者,平台转载素材出于传递更多信息,文章内容仅供参考与学习,切勿作为商业目的使用。如果侵害了您的合法权益,请您及时与我们联系,我们会在第一时间进行处理!我们尊重版权,也致力于保护版权,站搜网感谢您的分享!






